SSCP i MSSCP
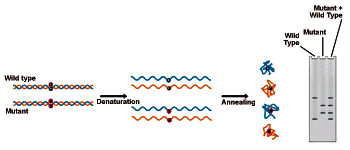
SSCP i MSSCP(z ang. single strand conformation polymorpfism), technika biologii molekularnej oparta na natywnym rozdziale elektroforetycznym kwasów nukleinowych, pozwalająca na różnicowanie fragmentów o tej samej długości ale odmiennej sekwencji. Metoda wykorzystywana przede wszystkim do wykrywania mutacji punktowych także w niejednorodnym materiale genetycznym. Zasada techniki SSCP polega na różnej ruchliwości elektroforetycznej jednoniciowych cząsteczek DNA w zależności od ich konformacji. Różna sekwencja fragmentu DNA skutkuje różnym stopniem skręcenia nici, który charakteryzuje określona zawada ruchu w żelu poliakrylamidowym. Aby wykonać rozdział SSCP, produkt PCR należy poddać denaturacji a następnie renaturacji. Czas trwania renaturacji jest na tyle krótki, iż nie jest możliwe odtworzenie podwójnych helis DNA a rozdzielone nici DNA skręcają się w obrębie jednej nici na skutek oddziaływania par A-T i G-C. Krytycznym parametrem stabilności powstałych konformerów DNA jest temperatura a jej ścisła kontrola ma kluczowe znaczenie dla jakości i powtarzalności wykonywanych rozdziałów. Ze względu na wpływ temperatury dla możliwości powstawanie i stabilizacji konformerów, dla zwiększenia prawdopodobieństwa wykrycia wszystkich możliwych konformerów, rozdział SSCP przeprowadza się trzykrotnie, w różnych temperaturach np. 35, 20 i 5 stopni C. Schematyczna ilustracja metody SSCP przedstawiona jest na rysunku 1.
Modyfikacją metody SSCP jest opracowana w Polsce metoda MSSCP (z ang. multitemperature single strand conformation polymorphism), w której, dzięki zastosowaniu dedykowanego urządzenia, możliwe jest przeprowadzenie jednego rozdziału elektroforetycznego w trzech temperaturach. Taki wariant metodyczny znacznie skraca czas wykonywanych analiz.
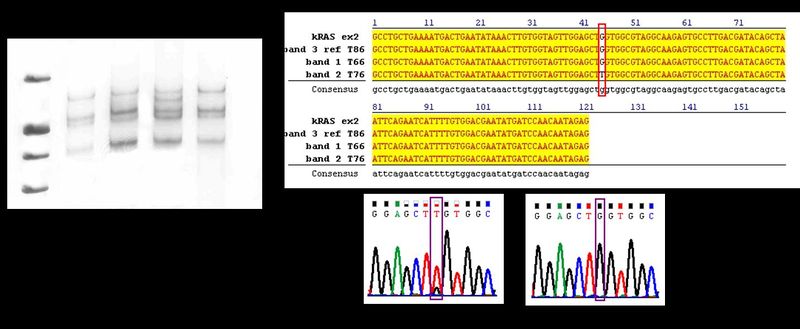
Optymalny rozdział SSCP/MSSCP pozwala na zróżnicowanie wariantu dzikiego (WT) i zmutowanego (MT), tak że są one widoczne jako prążki DNA na różnej wysokości żelu. Ważną zaletą tej metody jest także możliwość wykrycia nowych, nieznanych dotąd wariantów genetycznych. Każdy dodatkowy prążek może być bowiem reprezentacją nowego klonformeru DNA o nie opisywanej wcześniej zmianie w sekwencji nukleotydów. Pojedynczy prążek DNA można wyciąć z żelu, a zawarte w nim DNA odzyskać i zreamplifikować techniką PCR. Produkt PCR poddać następnie sekwencjonowaniu, które pozwala na potwierdzenie zmiany na poziomie sekwencji genu. Przykład wyniku tak przeprowadzonej analizy przedstawia rysunek 2. Zmiana w sekwencji dotyczy drugiego prążka od góry w rozdziale próbek T66 i T76.
Ważną aplikacją techniki SSCP/MSSCP jest jej wykorzystanie w analizie materiałów tkankowych, zawierających mieszaninę różnych komórek o różnych wariantach tego samego genu. Przykładem jest analiza biopsji guzów nowotworowych na przykład pod kątem markerów chemiooporności. Wiedząc, iż komórki stanowiące masę guza są zróżnicowane genetycznie, zastosowanie techniki SSCP/MSSCP pozawala na wykrycie wszystkich wariantów genu obecnych w różnych komórkach nowotworowych podczas jednej analizy.