(Utworzył nową stronę „==FoxO== Grupa białek FoxO należy do rodziny czynników transkrypcyjnych zawierających w swojej sekwencji konserwatywną domenę wiążącą się z DNA – Fox (...”) |
(→FoxO) |
||
| (Nie pokazano 1 pośredniej wersji utworzonej przez tego samego użytkownika) | |||
| Linia 6: | Linia 6: | ||
Enzymy komórkowe zaangażowane w modyfikacje potranslacyjne białek FoxO: | Enzymy komórkowe zaangażowane w modyfikacje potranslacyjne białek FoxO: | ||
| − | + | * fosforylacja: Akt/PKB, kinaza Ste20-like (MST1), c-Jun, ERK, JNK, p38, CDKs, AMPK, IkB. | |
| + | |||
[[Image:foxo_2__.jpg|thumb|center|800px| Miejsca fosforylacji białek FoxO1, FoxO3, FoxO4 w zależności od działającego enzymu przedstawia Tabela 1.]] | [[Image:foxo_2__.jpg|thumb|center|800px| Miejsca fosforylacji białek FoxO1, FoxO3, FoxO4 w zależności od działającego enzymu przedstawia Tabela 1.]] | ||
| − | + | ||
| − | + | * acetylacja: sirtuiny, deacetylazy histonów HDACs, acetylazy | |
| − | + | * ubikwitynacja: poliwubukwitynazy, ligazy, deubikwitynazy | |
| + | * metyzacja i glikozylacja: metyltransferaza argininy (PRMT1), O-glikozylacja, N-glikozylacja | ||
'''Piśmiennictwo:''' | '''Piśmiennictwo:''' | ||
Aktualna wersja na dzień 03:10, 26 lut 2015
FoxO
Grupa białek FoxO należy do rodziny czynników transkrypcyjnych zawierających w swojej sekwencji konserwatywną domenę wiążącą się z DNA – Fox (z ang. Forkhead box). Rodzina Fox obejmuje ponad 100 białek, które w zależności od podobieństwa sekwencji określone są od A do R. Występująca u ssaków grupa O obejmuje czynniki transkrypcyjne FoxO1, FoxO3a, FoxO4 i FoxO6. Białka FoxO są zaangażowane w wiele kluczowych procesów biologicznych, w tym areszt cyklu komórkowego, naprawę DNA, apoptozę, metabolizm glukozy, proces autofagii oraz starzenie się. Ze względu na udział białek FoxO w wielu procesach komórkowych, są one często związane z przebiegiem procesów starzenia, nowotworami, cukrzycą, niepłodnością, neurodegeneracją i dysfunkcją układu immunologicznego. Badania wykazały, iż zaburzenia aktywności FoxO prowadzą do rozwoju cukrzycy na tle zaburzeń wrażliwości komórek na działanie insuliny. W powiązaniu z chorobami nowotworowymi, czynniki FoxO są postulowane jako czynniki supresyjne. Utrata funkcji FoxO1, FoxO3 lub FoxO4 skutkuje rozwojem chłoniaka. Aby zrozumieć rolę czynników FoxO w patogenezie chorób należy wyjaśnić molekularny mechanizm ich oddziaływania oraz sposoby jego regulacji. Ogólnie ujmując, FoxO jest aktywowany przez różnego rodzaju modyfikacje potranslacyjne (PTM) takie jak: fosforylacja, acetylacja, ubikwitynacja, metyzacja czy glikozylacja. Modyfikacje FoxO zachodzą przy udziale specyficznych enzymów. Różnorodność modyfikacji FoxO umożliwia udział białka w wielu procesach komórkowych, zmianę jego lokalizacji, modyfikację oddziaływań z DNA czy wpływając na okres półtrwania białka. Schematyczna ilustracja możliwych modyfikacji białka FoxO1 jest przedstawiona na Rysunku 1.
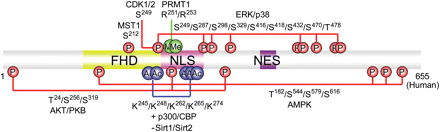
Enzymy komórkowe zaangażowane w modyfikacje potranslacyjne białek FoxO:
- fosforylacja: Akt/PKB, kinaza Ste20-like (MST1), c-Jun, ERK, JNK, p38, CDKs, AMPK, IkB.
- acetylacja: sirtuiny, deacetylazy histonów HDACs, acetylazy
- ubikwitynacja: poliwubukwitynazy, ligazy, deubikwitynazy
- metyzacja i glikozylacja: metyltransferaza argininy (PRMT1), O-glikozylacja, N-glikozylacja
Piśmiennictwo:
