TOPOIZOMERAZA TYPU I
Topoizomerazy typu I w procesie relaksacji DNA nacinają jedną nić i nie wymagają do tego hydrolizy ATP (oprócz odwrotnej gyrazy). W zależności od budowy, występowania i mechanizmu działania możemy podzielić je na trzy podgrupy: A, B i C [1]. Typ IA usuwa tylko negatywne superskręcenia. Typ IB usuwają zarówno pozytywne, jak i negatywne superskręcenia oraz ma nieco odmienny mechanizm relaksacji niż typ IA. Typ IC działa w sposób podobny do topoizomerazy IB, ale różni się od niej strukturą.
LUDZKA TOPOIZOMERAZA IB
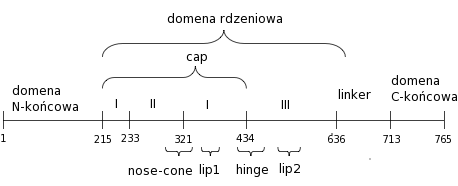
Przykładem topoizomerazy IB jest ludzka topoizmoeraza jądrowa. Składa się z 765 aminokwasów i ma masę 91 kDa. Można ją podzielić na 4 główne domeny: domenę N-końcową, domenę rdzeniową (rdzeń), linker i domenę C-końcową. Strukturalnie najbardziej interesująca jest domena rdzeniowa, gdyż z powodu swoich dużych rozmiarów i różnorodności funkcji jakie pełni, została podzielona jeszcze na trzy poddomeny (I, II, III - tzw. rdzeniowe), przy czym poddomeny I i II tworzą tzw. region cap. W domenie rdzeniowej można jeszcze wyróżnić kilka charakterystycznych rejonów: hinge, nose-cone oraz lip1 i lip2 (rys. 1 i 2).
Dotychczas poznano dwie funkcje topoizomerazy I: relaksacyjną (najbardziej znaną) i kinazową. Dla każdej z tych funkcji topoizomeraza przybiera odrębną konformację, a przełączanie pomiędzy konformacjami regulowane jest przez substraty tych reakcji. I tak: DNA blokuje aktywność kinazową topoizomerazy [3], a ATP [2,3], SF2/ASF [4,5] oraz substraty reakcji fosforylacji, blokują aktywność relaksacyjną. Konformacja jaką przybiera topoizomeraza w kompleksie z DNA jest dokładnie znana, gdyż istnieje wiele struktur krystalograficznych tego kompleksu w różnej rozdzielczości. Niestety, krystalograficzna struktura konformacji topoizomerazy w stanie niezwiązanym, do tej pory nie została rozwiązana, a wszystkie informacje dotyczące tej konformacji otrzymywane są z eksperymentów biologicznych i symulacji komputerowych.
AKTYWNOŚĆ RELAKSACYJNA
Najbardziej poznaną funkcją topoizomerazy I jest relaksacja DNA czyli usuwanie napięć torsyjnych powstałych podczas transkrypcji, replikacji, rekombinacji i kondensacji chromosomów. Konformacja topoizomerazy w kompleksie z DNA jest dobrze poznana, gdyż istnieją struktury krystalograficzne topoizomerazy I zarówno w formie związanej kowalencyjnie, jak i niekowalencyjnie. Nie są to jednak pełne struktury. Dotychczas wszystkie rozwiązane krystalograficznie struktury uzyskano dla topoizomerazy bez domeny N-końcowej, która nie jest jednak konieczna do relaksacji [6].
Topoizomeraza, w kompleksie z DNA, przybiera kształt zapięcia otaczającego DNA. Jedna część zapięcia jest tworzona przez region cap, a druga przez rdzeniową poddomenę III, domeną C-końcową i linker. Region łączący obie części zapięcia nazywany jest hinge i składa się z I i III poddomeny rdzeniowej (L429-K466). Po przeciwnej stronie DNA, patrząc wzdłuż osi helisy, niekowalencyjne zamknięcie zapięcia tworzą regiony cap-u i III poddomeny rdzeniowej, nazywane odpowiednio lip1 i lip2. Za zamknięcie zapięcia wokół DNA odpowiadają dwie pary oddziaływań: pierwsza to lizyna (K369 z lip1), która oddziałuje jonowo z kwasem glutaminianowym (E497 z lip2), druga to asparagina (N366 z lip1), która tworzy wiązanie wodorowe z kwasem asparaginowym (D500 z lip2). W regionie cap wyróżnia się rejon nose-cone (T303 - G339), tworzony przez dwie wysunięte helisy w kształcie litery V.
Proces relaksacji DNA przebiega w pięciu kolejnych etapach [7]:
- Topoizomeraza przyłącza się do DNA - zapięcie tworzone przez region cap z jednej strony, a rdzeniową poddomenę III i domenę C-końcową z drugiej, otwiera się. Zostało potwierdzone doświadczalnie, że kowalencyjne połączenie mostkami dwusiarczkowymi regionów lip1 i lip2 uniemożliwia wiązanie DNA.
- Zostaje przecięta jedna z nici DNA - przecięcie następuje w skutek ataku nukleofilowego tyrozyny 723 na fosfor 3' w DNA. Koniec 3’ przeciętej nici DNA zostaje kowalencyjnie przyłączony do topoizomerazy a drugi koniec pozostaje swobodny.
- DNA obraca się wokół nieprzeciętej nici. Prawdopodobnie ruch swobodnego końca DNA jest kontrolowany przez linker oraz rejon nose-cone [7] (choć pewne badania wskazują, że tylko przy usuwaniu negatywnego superskręcenia [8]). Istnieją symulacje komputerowe [8] sugerujące, że w zależności od tego, czy usuwane superskręcenie jest pozytywne czy negatywne, mechanizm takiej rotacji będzie inny. I tak przy usuwaniu pozytywnego superskręcenia rejony lip1 i lip2 topoizomerazy odsuwają się od siebie, a przy skręceniu negatywnym rejony lip1 i lip2 poruszają się nie znacznie, natomiast widoczne jest rozciąganie rejonu hinge.
- Ligacja pękniętej nici
- Oddysocjowanie topoizomerazy od DNA - prawdopodobnie zachodzi to w sposób odwrotny do przyłączania – czyli poprzez otwarcie zapięcia topoizomerazy.
AKTYWNOŚĆ KINAZOWA
Drugą ważną funkcją topoizomerazy IB jest aktywność kinazowa czyli fosforylacja białek z rodziny SR [9], odpowiedzialnych m.in. za regulacje splicingu, a także stabilności i transportu RNA [10,11]. Białka te składają się z jednej lub dwóch domen rozpoznających RNA (RRM – z ang.: RNA Recognition Motif) oraz słabo ustrukturyzowanej domeny C-końcowej, zwaną domena RS, bogatej w seryny i argininy, które mogą być fosforylowane. Najlepiej poznana jest interakcja topoizomerazy z alternatywnym czynnikiem splicingu SF2/ASF (SFRS1), który jest ważnym białkiem regulującym zarówno konstytutywny, jak i alternatywny splicing. W aktywność kinazową topoizomerazy zaangażowane są jej domeny: N-końcowa [12], rdzeniowa poddomena II [13] oraz domena C-końcowa [14]. Niestety konformacja jaką przyjmuje niezwiązana topoizomeraza nie jest znana, gdyż nie udało się dotychczas jej skrystalizować. Przyjmuje się jednak, że konformacja ta musi się różnić od konformacji topoizomerazy w kompleksie z DNA. Wiedza, dotycząca struktury kompleksu topoizomerazy z SF2, jaką dysponujemy pochodzi z doświadczeń biologii molekularnej oraz z symulacji komputerowych. I tak: aminokwasy K310 – M319 z poddomeny II oddziałują z domeną RRM1 białka SF2, domena N-końcowa topoizomerazy wiąże domenę RS białka SF2, a domena C-końcowa topoizomerazy wiąże ATP. Nie jest jednak wyjaśnione gdzie przyłącza się domena RRM2 białka SF2.
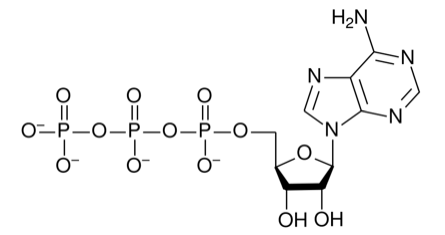
WIĄZANIE ATP
Związanie ATP przez jądrową topoizomerazę IB uruchamia mechanizm przełączenia aktywności topoizomerazy z relaksacyjnej na kinazową [3] a co za tym idzie powoduje zmianę jej konformacji. Samo zadokowanie ATP do topoizomerazy blokuje już wiązanie DNA do tej cząsteczki.
Nie jest do końca wyjaśnione gdzie znajduje się mięjsce wiązania ATP, choć pewne doświadczenia sugerują miejsce w domenie C-końcowej topoizomerazy [14], gdyż białko pozbawione tej domeny nie jest zdolne do wiązania ATP. Ponadto podczas trawienia topoizomerazy połączonej ze znakowanym izotopowo analogiem ATP (α-32P 8-azydoadenozyno-5'-trifosforanem), znakowane atomy znajdowano często związane z fragmentami domeny C-końcowej.
BIBLIOGRAFIA
- Baker N.M., Rajan R. Mondragón A. “Structural studies of type I topoisomerases.” Nucleic Acids Res 2009;37:693-701
- Castor F.J. & Kelly W.G. “ATP inhibits nuclear and mitochondrial type I topoisomerases from human leukemia cells.” Proc. Natl Acad. Sci. USA 1986;83:1680-1684.
- Chen H.J., Hwang J. “ Binding of ATP to human topoisomerase I resulting in an alteration of the conformation of the enzyme.” Eur J. Biochem 1999;265:367–375.
- Andersen F.F., Tange T.Ø., Sinnathamby T., Olsen J.R., Andersen K.E., Westergaard O., Kjems J., Knudsen B.R. “The RNA splicing factor ASF/SF2 inhibits human topoisomerase I mediated DNA relaxation.” J. Mol. Biol. 2002;322:677–686.
- Kowalska-Loth B., Girstun A., Piekiełko A., Staroń K. “SF2/ASF protein inhibits camptothecin-induced DNA cleavage by human topoisomerase I” Eur. J. Biochem. 2002;269:3504–3510.
- Alsner J., Svejstrup J.Q., Kjeldsen E., Sørensen B. S, Westergaard O. “Identification of an N-terminal domain of eukaryotic DNA topoisomerase I dispensable for catalytic activity but essential for in vivo function.” J. Biol. Chem. 1992;267:12408-12411.
- Stewart L., Redinbo M.R., Qiu X.Y., Hol W.G.J., Champoux J.J. “A model for the mechanism of human topoisomerase I.” Science 1998;279:1534–1541.
- Sari L., Andricioaei I. “Rotation of DNA around intact strand in human topoisomerase I implies distinct mechanisms for positive and negative supercoil relaxation” Nucleic Acids Res 2005;33:6621-6634.
- Rossi F., Labourier E., Forne´ T., Divita G., Derancourt J., Riou J.F., Antoine E., Cathala G., Brunel C., Tazi J. “Specific phosphorylation of SR proteins by mammalian DNA topoisomerase I.” Nature 1996;381:80–82.
- Lemaire R., Prasad J., Kashima T., Gustafson J., Manley J.L., Lafyatis R. “Stability of a PKCI-1-related mRNA is controlled by the splicing factor ASF/SF2: A novel function for SR proteins.” Genes Dev 2002;16:594–607.
- Huang Y., Steitz J.A. “Splicing factors SRp20 and 9G8 promote the nucleocytoplasmic export of mRNA.” Mol Cell 2001;7:899–905.
- Labourier E., Rossi F., Gallouzi I., Allemand E., Divita G., Tazi J. “Interaction between the N-terminal domain of human DNA topoisomerase I and the arginine-serine domain of its substrates determines phosphorylation of ASF/SF2 splicing factor.” Nucleic Acids Res 1998;26:2955–2962.
- Trzcińska-Daneluti A.M., Górecki A., Czubaty A., Kowalska-Loth B., Girstun A., Murawska M., Lesyng B., Staroń K. “RRM proteins interacting with the cap region of topoisomerase I.ę J Mol Biol 2007;369:1098–1112.
- Rossi F., Labourier E., Gallouzi I., Derancourt J., Allemand E., Divita G., Tazi J. “The C-terminal domain but not the tyrosine 723 of human DNA topoisomerase I active site contributes to kinase activity.” Nucleic Acids Res 1998;26:2963–2970.