(→RT-PCR- Reakcja Łańcuchowa Polimerazy z odwrotną transkrypcją) |
(→Literatura) |
||
| Linia 16: | Linia 16: | ||
W pierwszym wariancie całość reakcji zachodzi w jednej probówce co zapobiega kontaminacji próbki oraz ułatwia procesowanie. ''Two-step RT-PCR'' wymaga zastosowania dwóch probówek dla każdego z dwóch etapów. W pierwszym kroku przeprowadzana jest odwrotna transkrypcja całego RNA bądź poli(A)RNA na cDNA. W kolejnym etapie stosowane są już specyficzne primery, które pozwalają na amplifikację danej sekwencji cDNA. Ten rodzaj reakcji jest bardziej specyficzny. | W pierwszym wariancie całość reakcji zachodzi w jednej probówce co zapobiega kontaminacji próbki oraz ułatwia procesowanie. ''Two-step RT-PCR'' wymaga zastosowania dwóch probówek dla każdego z dwóch etapów. W pierwszym kroku przeprowadzana jest odwrotna transkrypcja całego RNA bądź poli(A)RNA na cDNA. W kolejnym etapie stosowane są już specyficzne primery, które pozwalają na amplifikację danej sekwencji cDNA. Ten rodzaj reakcji jest bardziej specyficzny. | ||
| − | + | '''Literatura''' | |
| − | http://www.e-biotechnologia.pl/Artykuly/rt-pcr | + | #http://www.e-biotechnologia.pl/Artykuly/rt-pcr |
| + | #http://www.lifetechnologies.com/pl/en/home/life-science/pcr/reverse-transcription/rt-pcr/one-two-step-rt-pcr.html | ||
| + | #McPherson M., Moller S. PCR. The BASICS. Second Edition (2006), Taylor & Francis Group. ISBN 0-4153-5547-8 | ||
| − | + | '''Ilustracja''' | |
| − | + | #http://www.abmgood.com/PCR/images/One-Step%20RT-PCRv2.jpg | |
| − | |||
| − | |||
| − | |||
| − | http://www.abmgood.com/PCR/images/One-Step%20RT-PCRv2.jpg | ||
Aktualna wersja na dzień 20:37, 11 lis 2014
RT-PCR- Reakcja Łańcuchowa Polimerazy z odwrotną transkrypcją
RT-PCR jest techniką bazującą na połączeniu reakcji PCR oraz odwrotnej transkrypcji. W odróżnieniu od PCR, pierwszym krokiem jest transkrypcja z cząsteczki RNA przez odwrotną transkryptazę (RNA-zależną polimerazę DNA) na cząsteczkę cDNA (ang. complementary DNA). Następnie przeprowadzana jest standardowa reakcja PCR (patrz: PCR). Zaletą tej metody jest bardzo duża specyficzność oraz czułość. Pozwala na określenie obecności ekspresji danego genu w komórce oraz poziomu jego ekspresji. Ponadto ułatwia pracę z RNA, które jest stosunkowo niestabilne i łatwo ulega degradacji.
Reakcję odwrotnej transkrypcji można przeprowadzać na całym RNA, bądź jedynie na mRNA. Ważnym elementem jest usunięcie genomowego DNA, które mogłoby być matrycą w standardowej reakcji PCR. W celu powielenia RNA często stosuje się oligo-T startery, które są komplementarne do końca 3’ poli-A mRNA. Najczęściej używane odwrotne transkryptazy to enzym wirusa ptasiej mieloblastomy (AMV) lub retrowirusa M-MLV. Zaletą transkryptazy AMV jest aktywność 3’-->5’ RNAzy H, która degraduje kompleksy RNA:DNA powstające podczas odwrotnej transkrypcji. W dalszej kolejności wykonywany jest PCR.
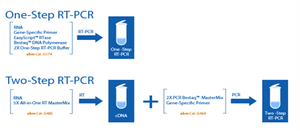
Weryfikowanie obecności produktu przeprowadza się w sposób podobny jak w standradowej rekacji PCR. Reakcję RT-PCR można przeprowadzić w dwojaki sposób (Rys. 1):
1. One step RT-PCR
2. Two-step RT-PCR
W pierwszym wariancie całość reakcji zachodzi w jednej probówce co zapobiega kontaminacji próbki oraz ułatwia procesowanie. Two-step RT-PCR wymaga zastosowania dwóch probówek dla każdego z dwóch etapów. W pierwszym kroku przeprowadzana jest odwrotna transkrypcja całego RNA bądź poli(A)RNA na cDNA. W kolejnym etapie stosowane są już specyficzne primery, które pozwalają na amplifikację danej sekwencji cDNA. Ten rodzaj reakcji jest bardziej specyficzny.
Literatura
- http://www.e-biotechnologia.pl/Artykuly/rt-pcr
- http://www.lifetechnologies.com/pl/en/home/life-science/pcr/reverse-transcription/rt-pcr/one-two-step-rt-pcr.html
- McPherson M., Moller S. PCR. The BASICS. Second Edition (2006), Taylor & Francis Group. ISBN 0-4153-5547-8
Ilustracja