(→Budowa:) |
|||
| (Nie pokazano 14 pośrednich wersji utworzonych przez tego samego użytkownika) | |||
| Linia 2: | Linia 2: | ||
INTEGRYNY – rodzina receptorow transbłonowych zaliczanych do białek adhezyjnych. Integryny tworzą połaczenia międzykomórkowe oraz połaczenia komórka-macierz zewnątrzkomorkowa (ECM). Pełnią również ważną funkcję strukturalną, poprzez wiązania z białkami cytoszkieletu oraz uczestniczą w transdukcji sygnału i odpowiedzi komórki na sygnały mikrośrodowiska. | INTEGRYNY – rodzina receptorow transbłonowych zaliczanych do białek adhezyjnych. Integryny tworzą połaczenia międzykomórkowe oraz połaczenia komórka-macierz zewnątrzkomorkowa (ECM). Pełnią również ważną funkcję strukturalną, poprzez wiązania z białkami cytoszkieletu oraz uczestniczą w transdukcji sygnału i odpowiedzi komórki na sygnały mikrośrodowiska. | ||
===Budowa:=== | ===Budowa:=== | ||
| − | Integryny są glikoproteinami. Każna z integryn jest hetorodimerem utworzonym z dwóch podjednostek: α i β. W genomie człowieka znanych jest 18 genów kodujących podjednostkę α oraz 8 genów kodujących podjednostkę β (tab.1). Dodatkowa zmienność jest zapewniana przez alternatywny splicing cząsteczek pre-mRNA kodujących podjednostki integrynowe. Kombinacja białek wchodzących w skład określonej integryny determinuje powinowactwo tego receptora do różnych ligandów (białek ECM). | + | Integryny są glikoproteinami. Każna z integryn jest hetorodimerem utworzonym z dwóch podjednostek: α i β. W genomie człowieka znanych jest 18 genów kodujących podjednostkę α oraz 8 genów kodujących podjednostkę β (tab.1). Dodatkowa zmienność jest zapewniana przez alternatywny splicing cząsteczek pre-mRNA kodujących podjednostki integrynowe. Kombinacja białek wchodzących w skład określonej integryny determinuje powinowactwo tego receptora do różnych ligandów (białek ECM). |
| − | Tabela 1. Geny kodujące podjednostki integryn. | + | {| class="wikitable" style="text-align:center" |
| − | + | |+ Tabela 1. Geny kodujące podjednostki integryn. | |
| − | + | ! colspan = 3 | PODJEDNOSTKA α | |
| − | + | ! colspan = 3 |PODJEDNOSTKA β | |
| − | ! PODJEDNOSTKA β | ||
|- | |- | ||
| Gen | | Gen | ||
| Białko | | Białko | ||
| − | | Alternatywne nazwy białka | + | | Alternatywne |
| + | nazwy białka | ||
| Gen | | Gen | ||
| Białko | | Białko | ||
| − | | Alternatywne nazwy białka | + | | Alternatywne |
| + | nazwy białka | ||
|- | |- | ||
| ITGA1 | | ITGA1 | ||
| + | | CD49a | ||
| + | | style="text-align:center" | VLA1 | ||
| + | | ITGB1 | ||
| + | | CD29 | ||
| + | | style="text-align:center" | MSK12, MDF2 | ||
| + | |- | ||
| + | | ITGA2 | ||
| + | | CD49b | ||
| + | | style="text-align:center" | VLA2 | ||
| + | | ITGB2 | ||
| + | | CD18 | ||
| + | | style="text-align:center" | LFA-1 | ||
| + | |- | ||
| + | | ITGA3 | ||
| + | | CD49c | ||
| + | | style="text-align:center" | VLA3 | ||
| + | | ITGB3 | ||
| + | | CD61 | ||
| + | | style="text-align:center" | GP3A | ||
| + | |- | ||
| + | | ITGA4 | ||
| + | | CD49d | ||
| + | | style="text-align:center" | VLA4 | ||
| + | | ITGB4 | ||
| + | | CD104 | ||
| + | | | ||
| + | |- | ||
| + | | ITGA5 | ||
| + | | CD49e | ||
| + | | style="text-align:center" | VLA5 | ||
| + | | ITGB5 | ||
| + | | ITGB5 | ||
| + | | style="text-align:center" | FLJ26658 | ||
| + | |- | ||
| + | | ITGA6 | ||
| + | | CD49f | ||
| + | | style="text-align:center" | VLA6 | ||
| + | | ITGB6 | ||
| + | | ITGB6 | ||
| + | | | ||
| + | |- | ||
| + | | ITGA7 | ||
| + | | ITGA7 | ||
| + | | style="text-align:center" | FLJ25220 | ||
| + | | ITGB7 | ||
| + | | ITGB7 | ||
| + | | | ||
| + | |- | ||
| + | | ITGA8 | ||
| + | | ITGA8 | ||
| + | | | ||
| + | | ITGB8 | ||
| + | | ITGB8 | ||
| + | | | ||
| + | |- | ||
| + | | ITGA9 | ||
| + | | ITGA9 | ||
| + | | style="text-align:center" | RLC | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGA10 | ||
| + | | ITGA10 | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGA11 | ||
| + | | ITGA11 | ||
| + | | style="text-align:center" | HsT18964 | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGAD | ||
| + | | CD11D | ||
| + | | style="text-align:center" | FLJ39841 | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGAE | ||
| + | | CD103 | ||
| + | | style="text-align:center" | HUMINAE | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGAL | ||
| + | | CD11a | ||
| + | | style="text-align:center" | LFA1A | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGAM | ||
| + | | CD11b | ||
| + | | style="text-align:center" | MAC-1 | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGAV | ||
| + | | CD51 | ||
| + | | style="text-align:center" | VNRA, MSK8 | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGA2B | ||
| + | | CD41 | ||
| + | | style="text-align:center" | GPIIb | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | |- | ||
| + | | ITGAX | ||
| + | | CD11c | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
|} | |} | ||
| + | |||
===Funkcja:=== | ===Funkcja:=== | ||
# Tworzenie połaczeń adhezyjnych. | # Tworzenie połaczeń adhezyjnych. | ||
Aktualna wersja na dzień 09:46, 3 paź 2014
Integryny
INTEGRYNY – rodzina receptorow transbłonowych zaliczanych do białek adhezyjnych. Integryny tworzą połaczenia międzykomórkowe oraz połaczenia komórka-macierz zewnątrzkomorkowa (ECM). Pełnią również ważną funkcję strukturalną, poprzez wiązania z białkami cytoszkieletu oraz uczestniczą w transdukcji sygnału i odpowiedzi komórki na sygnały mikrośrodowiska.
Budowa:
Integryny są glikoproteinami. Każna z integryn jest hetorodimerem utworzonym z dwóch podjednostek: α i β. W genomie człowieka znanych jest 18 genów kodujących podjednostkę α oraz 8 genów kodujących podjednostkę β (tab.1). Dodatkowa zmienność jest zapewniana przez alternatywny splicing cząsteczek pre-mRNA kodujących podjednostki integrynowe. Kombinacja białek wchodzących w skład określonej integryny determinuje powinowactwo tego receptora do różnych ligandów (białek ECM).
| PODJEDNOSTKA α | PODJEDNOSTKA β | ||||
|---|---|---|---|---|---|
| Gen | Białko | Alternatywne
nazwy białka |
Gen | Białko | Alternatywne
nazwy białka |
| ITGA1 | CD49a | VLA1 | ITGB1 | CD29 | MSK12, MDF2 |
| ITGA2 | CD49b | VLA2 | ITGB2 | CD18 | LFA-1 |
| ITGA3 | CD49c | VLA3 | ITGB3 | CD61 | GP3A |
| ITGA4 | CD49d | VLA4 | ITGB4 | CD104 | |
| ITGA5 | CD49e | VLA5 | ITGB5 | ITGB5 | FLJ26658 |
| ITGA6 | CD49f | VLA6 | ITGB6 | ITGB6 | |
| ITGA7 | ITGA7 | FLJ25220 | ITGB7 | ITGB7 | |
| ITGA8 | ITGA8 | ITGB8 | ITGB8 | ||
| ITGA9 | ITGA9 | RLC | |||
| ITGA10 | ITGA10 | ||||
| ITGA11 | ITGA11 | HsT18964 | |||
| ITGAD | CD11D | FLJ39841 | |||
| ITGAE | CD103 | HUMINAE | |||
| ITGAL | CD11a | LFA1A | |||
| ITGAM | CD11b | MAC-1 | |||
| ITGAV | CD51 | VNRA, MSK8 | |||
| ITGA2B | CD41 | GPIIb | |||
| ITGAX | CD11c | ||||
Funkcja:
- Tworzenie połaczeń adhezyjnych.
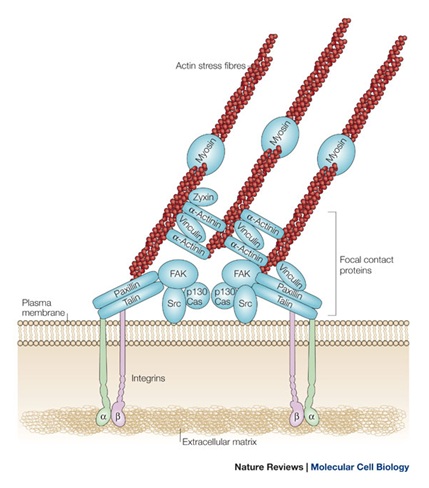
Integryny umożliwiają przytwierdzenie komórki do podłoża poprzez tworzenie połączeń zwierających komórka-macierz zewnątrzkomórkowa (ECM). W połączeniu typu przyczep ogniskowy (ang. focal adhesion) zewnątrzkomórkowa domena integryny wiąże się z białkami ECM (np. fibronektyną), a część cytoplazmatyczna pozostaje związana z filamentami aktynowymi poprzez białka kotwiczące: talinę, paksylinę, winkulinę i α-aktyninę (ryc. 1) [1, 2]. Innym typem integrynowych połaczeń zwierających są hemidesmosomy wiążące komórkę do błony podstawnej. Hemidesmosomy są związane z cytoszkieletem komórki (filamentami pośrednimi) za pomocą pochodnych desmoplakiny [1].
- Transdukcja sygnału
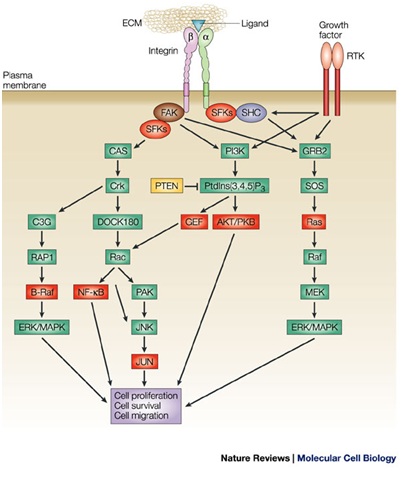
Integryny są receptorami błonowymi przenoszącymi informacje na temat struktury oraz składu otoczenia komórki. Ligandami dla integryn są: fibronektyna, witronektyna, kolagen oraz laminina. Integryny oraz błonowe receptory kinaz tyrozynowych (RTK - receptor tyrosine kinase) determinują odpowiedź komórki na poziomie: prolifracji, migracji lub apoptozy (ryc.2) [3].
- Udział w procesie migracji komórki.
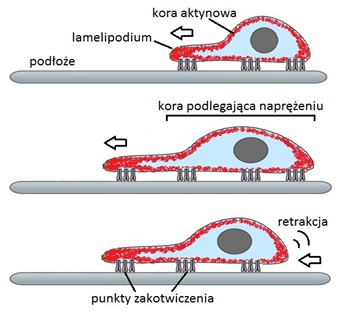
Integryny tworzą punkty zakotwiczenia komórki do macierzy zewnątrzkomórkowej charakterystyczne dla procesu migracji komórki [4]. Wypustka cytoplazmy zwrócona w kierunku ruchu komórki (lamelipodium) tworzy cienkie palczaste wypustki (filopodia), które łączą się z podłożem za pomocą połaczeń integrynowych. Powoduje to naprężenia w cytoszkielecie i oderwanie się od podloża tylnej części komórki oraz depolimeryzacji filamentów aktynowych (retrakcja) (Ryc.3).
Brak wiązań receptorów integrynowych z ligandami indukuje szlak sygnałowy prowadzący do apoptozy (tzw. anoikis).
Literatura
- Alberts B, Johnson A, Lewis J, et al. „Molecular Biology of the Cell. 4th edition.” New York: Garland Science; 2002.
- Mitra SK, Hanson DA, Schlaepfer DD. „Focal adhesion kinase: in command and control of cell motility.” Nat Rev Mol Cell Biol. 2005 Jan;6(1):56-68. Review. 15688067.
- Guo W, Giancotti FG. Integrin signalling during tumour progression. Nat Rev Mol Cell Biol. 2004 Oct;5(10):816-26. 15459662.
- Ngalim SH, Magenau A, Le Saux G, Gooding JJ, Gaus K. „How do cells make decisions: engineering micro- and nanoenvironments for cell migration.” J Oncol. 20652046.